Che cos'è GLM in R?
I modelli lineari generalizzati sono un sottoinsieme di modelli di regressione lineare e supportano efficacemente le distribuzioni non normali. Per supportare ciò si consiglia di utilizzare la funzione glm (). GLM funziona bene con una variabile quando la varianza non è costante e distribuita normalmente. È stata definita una funzione di collegamento per trasformare la variabile di risposta in modo che si adatti al modello appropriato. Un modello LM viene eseguito sia con la famiglia che con la formula. Il modello GLM ha tre componenti chiave chiamate random (probabilità), sistematica (predittore lineare), componente link (per la funzione logit). Il vantaggio di usare glm è che hanno la flessibilità del modello, non c'è bisogno di una varianza costante e questo modello si adatta alla stima della massima verosimiglianza e ai suoi rapporti. In questo argomento, impareremo a conoscere GLM in R.
Funzione GLM
Sintassi: glm (formula, famiglia, dati, pesi, sottoinsieme, Start = null, modello = TRUE, metodo = ””…)
Qui i tipi di famiglia (include i tipi di modello) includono binomiale, Poisson, gaussiano, gamma, quasi. Ogni distribuzione esegue un uso diverso e può essere utilizzata in classificazione e previsione. E quando il modello è gaussiano, la risposta dovrebbe essere un numero intero reale.
E quando il modello è binomiale, la risposta dovrebbe essere una classe con valori binari.
E quando il modello è Poisson, la risposta dovrebbe essere non negativa con un valore numerico.
E quando il modello è gamma, la risposta dovrebbe essere un valore numerico positivo.
glm.fit () - Per adattarsi a un modello
Lrfit () - indica adattamento della regressione logistica.
update () - aiuta ad aggiornare un modello.
anova () - è un test opzionale.
Come creare GLM in R?
Qui vedremo come creare un modello lineare generalizzato facile con dati binari usando la funzione glm (). E proseguendo con il set di dati Alberi.
Esempi
// Importazione di una librerialibrary(dplyr)
glimpse(trees)

Per vedere i valori categorici sono assegnati dei fattori.
levels(factor(trees$Girth))
// Verifica delle variabili continue
library(dplyr)
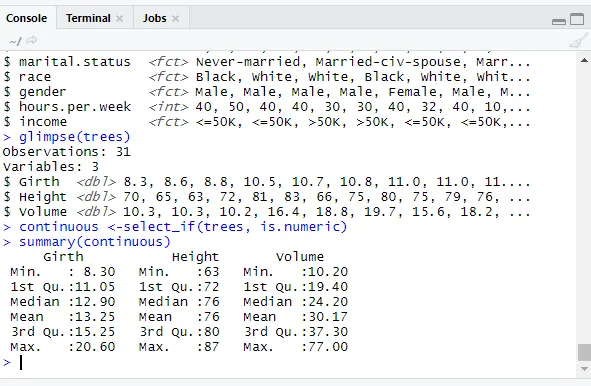
continuous <-select_if(trees, is.numeric)
summary(continuous)
// Incluso set di dati dell'albero nella ricerca R Pathattach (alberi)
x<-glm(Volume~Height+Girth)
x
Produzione:
| Chiama: glm (formula = Volume ~ Altezza + Circonferenza)
coefficienti: (Intercetta) Altezza circonferenza -57.9877 0.3393 4.7082 Gradi di libertà: 30 totali (cioè Null); 28 Residuo Deviazione nulla: 8106 Devianza residua: 421, 9 AIC: 176, 9 |
summary(x)
| Chiamata:
glm (formula = Volume ~ Altezza + Circonferenza) Residui di devianza: Min 1Q Mediana 3Q Max -6, 4065 -2, 6493 -0, 2876 2, 2003 8, 4847 coefficienti: Stima std. Valore t errore Pr (> | t |) (Intercetta) -57.9877 8.6382 -6.713 2.75e-07 *** Altezza 0.3393 0.1302 2.607 0.0145 * Sottopancia 4.7082 0.2643 17.816 <2e-16 *** - Signif. codici: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 '' 1 (Parametro di dispersione per la famiglia gaussiana considerato essere 15.06862) Deviazione nulla: 8106.08 su 30 gradi di libertà Devianza residua: 421, 92 su 28 gradi di libertà AIC: 176.91 Numero di iterazioni del punteggio Fisher: 2 |
L'output della funzione di riepilogo fornisce le chiamate, i coefficienti e i residui. La risposta sopra riportata dimostra che sia l'altezza che la circonferenza non sono significative in quanto la loro probabilità è inferiore a 0, 5. E ci sono due varianti di devianza chiamate null e residual. Infine, il punteggio del pescatore è un algoritmo che risolve i problemi di massima probabilità. Con il binomio, la risposta è un vettore o una matrice. cbind () è usato per legare i vettori di colonna in una matrice. E per ottenere le informazioni dettagliate del riepilogo di adattamento viene utilizzato.
Per eseguire il test Hood come viene eseguito il seguente codice.
step(x, test="LRT")
Start: AIC=176.91
Volume ~ Height + Girth
Df Deviance AIC scaled dev. Pr(>Chi)
421.9 176.91
- Height 1 524.3 181.65 6.735 0.009455 **
- Girth 1 5204.9 252.80 77.889 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Call: glm(formula = Volume ~ Height + Girth)
Coefficients:
(Intercept) Height Girth
-57.9877 0.3393 4.7082
Degrees of Freedom: 30 Total (ie Null); 28 Residual
Null Deviance: 8106
Residual Deviance: 421.9 AIC: 176.9
Vestibilità modello
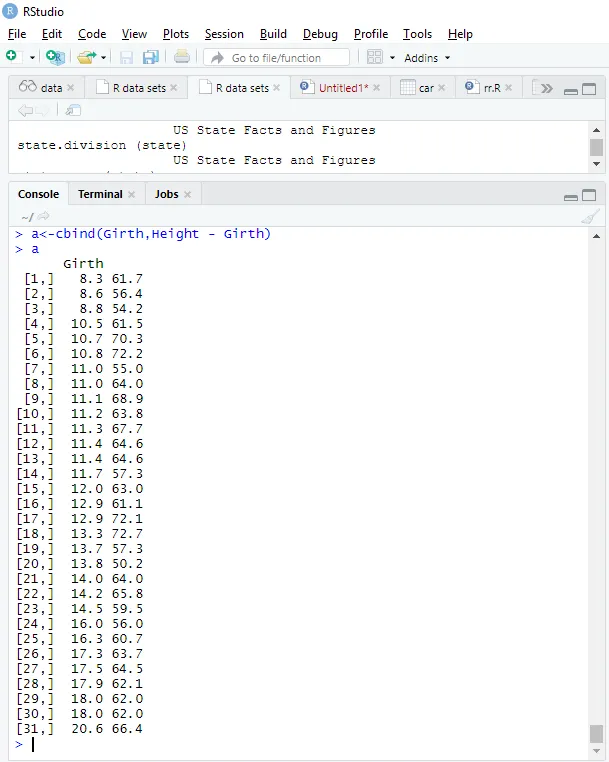
a<-cbind(Height, Girth - Height)
> a
riassunto (alberi)
Girth Height Volume
Min. : 8.30 Min. :63 Min. :10.20
1st Qu.:11.05 1st Qu.:72 1st Qu.:19.40
Median :12.90 Median :76 Median :24.20
Mean :13.25 Mean :76 Mean :30.17
3rd Qu.:15.25 3rd Qu.:80 3rd Qu.:37.30
Max. :20.60 Max. :87 Max. :77.00
Per ottenere la deviazione standard appropriata
apply(trees, sd)
Girth Height Volume
3.138139 6.371813 16.437846
predict <- predict(logit, data_test, type = 'response')
Successivamente, ci riferiamo alla variabile di risposta al conteggio per modellare un buon adattamento di risposta. Per calcolare questo, useremo il set di dati USAccDeath.
Inseriamo i seguenti frammenti nella console R e vediamo come vengono eseguiti il conteggio degli anni e il quadrato degli anni.
data("USAccDeaths")
force(USAccDeaths)
// Analizzare l'anno 1973-1978.
disc <- data.frame(count=as.numeric(USAccDeaths), year=seq(0, (length(USAccDeaths)-1), 1)))
yearSqr=disc$year^2
a1 <- glm(count~year+yearSqr, family="poisson", data=disc)
summary(a1)
| Chiamata:
glm (formula = count ~ year + yearSqr, family = “poisson”, data = disc) Residui di devianza: Min 1Q Mediana 3Q Max -22.4344 -6.4401 -0.0981 6.0508 21.4578 coefficienti: Stima std. Valore z errore Pr (> | z |) (Intercettazione) 9.187e + 00 3.557e-03 2582.49 <2e-16 *** anno -7.207e-03 2.354e-04 -30.62 <2e-16 *** annoSqr 8.841e-05 3.221e-06 27.45 <2e-16 *** - Signif. codici: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 '' 1 (Il parametro di dispersione per la famiglia Poisson è considerato 1) Deviazione nulla: 7357, 4 su 71 gradi di libertà Devianza residua: 6358, 0 su 69 gradi di libertà AIC: 7149, 8 Numero di iterazioni del punteggio Fisher: 4 |
Per verificare il miglior adattamento del modello, è possibile utilizzare il seguente comando per trovare
i residui per la prova. Dal risultato seguente il valore è 0.
1 - pchisq(deviance(a1), df.residual(a1))
Utilizzo della famiglia QuasiPoisson per la maggiore varianza nei dati forniti
a2 <- glm(count~year+yearSqr, family="quasipoisson", data=disc)
summary(a2)
| Chiamata:
glm (formula = count ~ year + yearSqr, family = “quasipoisson”, dati = disco) Residui di devianza: Min 1Q Mediana 3Q Max -22.4344 -6.4401 -0.0981 6.0508 21.4578 coefficienti: Stima std. Valore t errore Pr (> | t |) (Intercettazione) 9.187e + 00 3.417e-02 268.822 <2e-16 *** anno -7.207e-03 2.261e-03 -3.188 0.00216 ** annoSqr 8.841e-05 3.095e-05 2.857 0.00565 ** - (Parametro di dispersione per famiglia quasipoisson considerato 92.28857) Deviazione nulla: 7357, 4 su 71 gradi di libertà Devianza residua: 6358, 0 su 69 gradi di libertà AIC: NA Numero di iterazioni del punteggio Fisher: 4 |
Il confronto tra Poisson e il valore AIC binomiale differisce in modo significativo. Possono essere analizzati con precisione e rapporto di richiamo. Il prossimo passo è verificare che la varianza dei residui sia proporzionale alla media. Quindi possiamo tracciare usando la libreria ROCR per migliorare il modello.
Conclusione
Pertanto, ci siamo concentrati su un modello speciale chiamato modello lineare generalizzato che aiuta a focalizzare e stimare i parametri del modello. È principalmente il potenziale per una variabile di risposta continua. E abbiamo visto come glm si adatta a pacchetti integrati R. Sono gli approcci più popolari per misurare i dati di conteggio e uno strumento robusto per le tecniche di classificazione utilizzate da uno scienziato di dati. Il linguaggio R, ovviamente, aiuta a svolgere complicate funzioni matematiche
Articoli consigliati
Questa è una guida a GLM in R. Qui discutiamo della funzione GLM e di come creare GLM in R con esempi e output di set di dati dell'albero. Puoi anche leggere il seguente articolo per saperne di più -
- R Linguaggio di programmazione
- Architettura dei big data
- Regressione logistica in R
- Lavori di analisi dei Big Data
- Regressione di Poisson in R | Implementazione della regressione di Poisson